Gene Cloning
實驗室常常會聽到 “cloning” 這個詞,老師偶爾會請你 “cloning” 一段基因,到底 “cloning” 是什麼意思呢?Cloning 指的是從一團多樣的 DNA 片段混合物中,挑選出特定的序列片段,黏接到「載體」上。將「載體」送到研究目標中 (如細菌和細胞),並在目標中大量表現,來研究此特定序列對於目標的影響。
傳統上通常會使用聚合酶連鎖反應 (polymerase chain reaction, PCR),「夾」出特定目標片段,此步驟有時需要一定的運氣,需要嘗試不同的「夾子」和反應條件。而近代科技的進步則推出另外一種方法——直接使用化學的方式全序列合成,不過此方法非常昂貴。
Cloning 大致可以分成四步:
此步驟為聚合酶連鎖反應的前置作業,必須先獲得含有目標片段的混合物。可以是染色體 DNA 或是由 RNA 反轉錄形成的 cDNA (complementary DNA),而抽取的來源可以是細菌、細胞或是動植物組織。同一種 DNA 類型 (染色體或 cDNA) 抽取原理差不多,不同來源可能會有些微差異,差異主要是破細胞的部分,比如說組織必須先均質化,去掉一些組織間質,而細菌有細胞壁,相比於細胞而言較難破壞。現在為了方便科學家的研究,都有對應的套組。最後得到的 DNA 在下個步驟中稱為「模板」,用來當作聚合酶連鎖反應的反應物。
破壞細胞的方式可分成物理法和化學法,化學法是直接使用 lysis buffer溶解細胞外層,物理法則有超音波、擠壓法、液態氮凍融法等。之後抽取過程中,由於染色體 DNA 片段較長,因此需要小心避免斷裂。傳統上,染色體 DNA 抽取時,會使用 proteinase K 分解細胞中蛋白質,再使用高鹽溶液沉澱蛋白質,透過離心去掉雜質,最後使用異丙醇沉澱出 DNA。需要特別注意的是,染色體 DNA 因為分子量巨大,所以在回溶時需要提高溫度或是延長時間,建議55oC 一小時或室溫過夜。而ACE Biolabs 也有提供管柱的方法 (ACExtract DNA Isolation Kit(from Cell/Tissue, ACE Biolabs CE1006),可以節省沉澱和回溶的時間。為了確認染色體 DNA 的完整性,會使用瓊脂糖膠體電泳確認 (請見下方「二.聚合酶連鎖反應:「夾」出特定目標片段——E.聚合酶連鎖反應確認」),樣本應該要位於 10kb 以上,並且沒有明顯的小片段脫尾。
而 RNA 雖然是多條小片段,但 RNA 本身較脆弱容易降解,所以實驗中需要保持乾淨。傳統上,RNA 抽取是使用有機溶劑變性蛋白質,透過離心分成有機層和水層,DNA 和蛋白質會留在有機層,而 RNA 則在水層,最後使用異丙醇沉澱出 RNA。而ACE Biolabs 也有提供管柱的方法 (ACExtract Total RNA Extraction Reagent, ACE Biolabs CE1003),可以節省沉澱和回溶的時間,以及增加安全性和避免有機溶劑汙染。為了確認 RNA 的完整性 (沒有降解),會使用瓊脂糖膠體電泳確認 (請見下方「二.聚合酶連鎖反應:「夾」出特定目標片段——E.聚合酶連鎖反應確認」),主要是看在 RNA 中最多的核醣體 RNA (rRNA),都會有三條明顯訊號 (真核是5/18/28S,細菌是5/16/23S)。如果是使用有機溶劑抽取的話,還要特別注意260/230吸光值比例必須要大於2,小於2則代表有機溶劑汙染,會影響後續的反應。純化的 RNA 還需要經過反轉錄成 cDNA,可以大幅度地增加保存的穩定性,ACE Biolabs 提供5X ACE Script II 1st strand cDNA RT SuperMix (+gDNA wiper) (ACE Biolabs, EP2015)。
聚合酶連鎖反應可以說是 cloning 中最重要的步驟,也是生物裡非常特別的機制。大自然中幾乎所有生物的遺傳物質都是 DNA,而生物能夠一代代地繁衍的其中一個關鍵就是「傳遞遺傳物質」。經過科學家們的努力研究,才發現 DNA 複製的機制為「半保留複製」。DNA 為雙股結構,透過鹼基配對的方式 (ATCG) 形成氫鍵穩定結構。DNA 複製時,雙股分開來,兩股都當作模板,透過鹼基配對的方式接上新的鹼基,做出配對股 (complementary strand)。新形成的兩條雙股 DNA 都同時有一股舊的和一股新的 DNA,因此稱作「半保留複製」。
實際上細胞內 DNA 複製非常複雜,需要多種酵素的參與,而科學家們就是將這個反應搬到試管內,盡量地減少酵素的使用,而使用其他方式代替。其中最重要並且無可替代的就是聚合酶 (DNA polymerase),負責辨認和配對鹼基。聚合酶反應中還需要引子、dNTP 和 Mg2+ 以及適合的反應鹽離子液。dNTP 就是四種鹼基的合稱,提供複製時的原料和能量,Mg2+ 則是聚合酶的輔因子,幫助穩定 DNA 結構。
最關鍵的就是引子,引子為反應時的定位點,聚合酶會先辨認到引子,往下進行反應,因此可以透過設計引子序列「夾出」目標片段。引子其實就是短片段的 DNA,會設計成和配對股完全相同的序列。由於聚合酶反應有順序性,做出來的配對股必須由 DNA 5’端到3’端,因此加入符合目標片段前後序列的引子,再加上限縮反應的時間,可以順利地放大目標片段。網路上有很多免費的引子設計軟體,可以自己調整長度、GC content、Tm、self-annealing、pair-annealing。
引子的好壞除了本身的特性之外,最重要的是專一性,假設引子長度為30b.p.,代表其專一性應該為 (1/4)^30。但是實際上由於模板 DNA 非常龐大,加上裡面的反應並不像預期地如此簡單,這些分子很容易發生交互作用。因此,有時候需要試驗多組引子才能成功放大出目標片段。
除了上述材料之外,還需要三步驟溫度的改變:變性 denaturation、黏合 annealing、擴增 extension,而以舊的模板做出來的配對股又可以在下一次反應中當作模板,因此透過三個步驟的不斷循環,可以指數地放大出目標片段,所以稱作「連鎖反應」 (圖一)。通常循環數會設在25-35個循環,過少的話放大的目標片段太少,過多的話容易出現非專一性訊號。
初始變性階段:95oC 5-10分鐘
25-35個循環:變形95oC 30秒、黏合55-65oC 30秒、擴增72oC
最終擴增階段:72oC 5-10分鐘
早期只能透過不同的水浴槽,手動改變反應溫度。後來才設計出自動控溫的機器 (PCR machine),可以設定各種程式,多塊控溫晶片的設計使得我們可以一次試驗不同的黏合溫度,就連升降溫的速率都可以控制。而有時因為目標片段過長,而有較長的反應時間,機器也可以設定在4oC 方便保存。
目前市面上販售的聚合酶試劑中通常有聚合酶、反應液 (2-10倍)、Mg2+、dNTP,有些廠商會將 Mg2+ 併到反應液中,節省操作步驟。而 DNA 模板和引子由實驗者自己準備,DNA 引子需要請其他廠商合成。操作時要注意,聚合酶是生物酵素,需要低溫取用 (通常於-20oC 保存時為液體,可直接取用);而 dNTP 容易因為反覆凍融失去活性,可於第一次使用時分裝。ACE Biolabs 提供的 ACE Super-Fidelity DNA PCR kit (ACE Biolabs, EP1005),具有高效率、高連續性以及校正的能力,可以更快更精確地放大出目標片段。
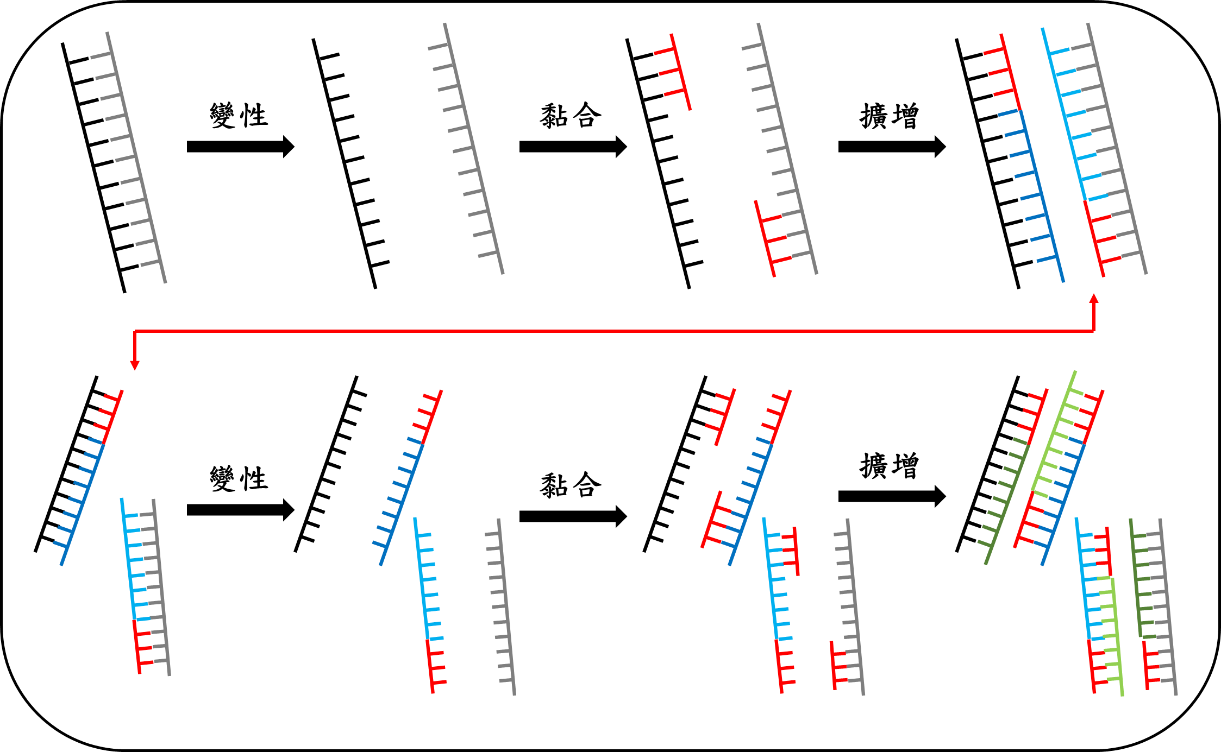
圖一:聚合酶連鎖反應示意圖,第一次做出來的配對股可以在第二次反應中當作模板,再製作出配對股。
聚合酶有四大特性,專一性 (specificity)、熱穩定性 (thermostability)、正確性 (fidelity) 和連續性 (processivity)。
聚合酶的專一性決定於酵素本身和環境溫度,酵素本身的構形會專一性地配對鹼基,而環境溫度則決定酵素活性和構形。酵素都有最適合作用的溫度,即使處在其他溫度,聚合酶依然有一定的活性,但是卻會導致專一性大幅度下降,而錯誤地放大出非目標片段。最簡單的解決方法是冰上操作,在機器溫度到達變性溫度前,配好的反應液必須置於冰上,降低酵素活性。
另一個方法則是 “hot start”,加入特殊的抗體抑制聚合酶,只有當溫度超過90oC 時,抗體才會失活及降解,聚合酶才能作用,大幅度地提高專一性。
就如同上述所提過的,在變性高溫下,一般的生物酵素都會永久地失去活性,直到後來科學家們發現了熱嗜菌,這種生物的酵素適合溫度位於50-80oC,並且可以耐受高溫而保持活性。
聚合酶除了透過蛋白質構形來決定下一個鹼基,還有另外一個機制可以提高正確性。聚合酶除了有「5’到3’複製 DNA」的能力之外,還有「3’到5’剪切 DNA (3’ to 5’ exonuclease 外切酶 )」的特殊能力。酵素會「檢查」做過的部分,發現錯誤後就會停頓,去除掉錯誤的部分,再重新黏上正確的鹼基,這項能力也被稱作「proofreading」。聚合酶配對鹼基的錯誤率大約是1/10^5,也就是每100,000鹼基中會有一個錯誤,而 proofreading 的特性可以降低100倍,使錯誤率下降至1/10^7。
連續性的意思是代表聚合酶一次作用的連續長度。在生物體內,聚合酶在 DNA 上移動時,有其他多種蛋白質協助「聚合酶—DNA」異構物的穩定。而在試管內反應時,缺乏那些蛋白質的幫忙,聚合酶很容易「鬆脫」,使得無法做出目標片段全長。因此,如果目標片段較長,則必須選擇高連續性的聚合酶。ACE Super-Fidelity DNA PCR kit (ACE Biolabs, EP1005) 即可用來針對長片段目標,並且能夠抵抗常見的抑制物 (heparin, xylan, humic acid 等)。
完成聚合酶連鎖反應後,通常會使用瓊脂糖膠體電泳 (agarose gel electrophoresis) 分析。瓊脂糖膠體電泳是用來分析大分子核酸 (DNA 和 RNA) 的方法。瓊脂糖是由石花菜屬和江蘺屬藻類中萃取出來的多糖類,於室溫時不溶於水,必須透過加熱到90-95oC 才能溶解。而在緩慢冷卻到室溫時,瓊脂糖會緩慢地形成有序的網狀結構。而在製備膠體時,必須確認瓊脂糖白色粉末完全溶解,冷卻溶解的膠體至50-60oC 後,倒入鑄膠槽,並快速地插入齒梳,待膠體冷卻後,即可留下樣本槽。ACE Biolabs 提供 Agarose LE (ACE Biolabs C3016)。
核酸本身帶負電,在電場作用下,會由負極往正極移動。核酸長度越長,越不容易通過網子,移動地越慢,因此瓊脂糖膠體可以用來分離不同長度的核酸。瓊脂糖濃度越大,網子間的距離越小,即膠體的孔隙越小,可以用來分離較小差別的核酸。通常1%瓊脂糖膠體可以用來區分0.5-10k.b. 核酸,解析度大約是300-1000b.p.,長度越長則需要較大的差異才能分辨出來。而2%瓊脂糖膠體則用來區分100-1000b.p. 以下核酸,解析度大約是50-100b.p.。瓊脂糖膠體的解析極限就是100b.p. 左右,即使提高濃度也無法有效增加,更小片段的長度需要用聚丙烯醯胺凝膠電泳 (SDS-PAGE) 才能分辨 (需要特殊的緩衝液)。
配置膠體時需要特別注意不能使用二次水,而必須使用電泳緩衝液,以確保電泳環境一致。而瓊脂糖膠體電泳使用的緩衝液可以使用 TAE (Tris-acetate-EDTA) 或 TBE (Tris-borate-EDTA),兩者都是緩衝液,只不過使用不同的酸配置 (acetate 或 borate)。TAE膠體中 DNA 回收率較高,而且價格比較便宜,並且可以配製成50倍稀釋使用;然而解析度較低,容易產熱。TBE 解析度較高,比較不容易產熱,但是價格較貴,而且由於 boric acid 容易沉澱,只能配成5倍溶液。
另外,還有三個東西要添加:DNA 染劑、DNA loading dye和 DNA marker。
DNA 分子本身沒有螢光特性,因此必須要加入 DNA 染劑,方便我們觀察 DNA。早期使用的 EtBr 是透過平板的結構嵌入 DNA 雙股螺旋,在紫外光激發下,EtBr 分子和 DNA 作用後會放出強烈的橙紅色螢光。但是也因為嵌入 DNA 的特性,使得 EtBr 具有高突變性和致癌性,使用時要特別注意。近代研發的 DNA 安全染劑雖然價格較貴,但是大幅度地降低危險,並且使用藍光激發 (黃綠色螢光),也較為安全。ACE Biolabs 提供 DNA Safeview Dye (ACE Biolabs ER1012)。
依據 DNA 染劑使用時機可分成內染和外染,外染指的是電泳完成後,將膠體浸泡到含有 DNA 染劑的二次水中 (通常是1/1000-1/2000稀釋倍率),等待約5-10分鐘,再取出膠體至二次水中「退染」。要注意染缸和退染缸要定期更換,因為 DNA 染劑容易光解,而退染用的二次水會因為使用次數太多,而有太多的染劑使退染效果降低。外染的優點在於製備膠體比較容易,染膠區和電泳區可以分開,最後不會有不均勻的螢光結果。內染指的是製備瓊脂糖膠體時,就一起加入染劑 (通常是1/10,000-1/20,000倍率稀釋),因此形成的凝固膠體即含有 DNA 染劑。進行電泳時,DNA 染劑會附著在 DNA 上。優點在於解析度較高和節省時間,但因為螢光染劑本身帶正電,會隨電場一起移動,因此會出現膠體上面較亮下面較暗的結果。
包含甘油 (glycerol)、EDTA 以及染劑 (通常是 bromophenol blue 和 xylene cyanol)。
DNA 電泳中,只能判斷長度的相對關係,無法了解絕對的長度,因此需要另外的標準。而 DNA marker 就像一把尺,提供數字大小的刻度。裡頭包含不同長度的 DNA,通常市面上有兩種,一種稱作「1kb marker」,範圍250 b.p.-10 k.b.,主要搭配 1k.b. 以上的目標片段使用;另一種稱為「100b.p. marker」,範圍100 b.p.-3 k.b.,主要搭配100-1000 b.p. 目標片段使用。除了依據目標片段長度,來決定使用的 DNA marker,同時也會搭配到瓊脂糖膠體的濃度,1% 膠體搭配「1kb marker」,可以分辨出1-10 k.b. 片段,1 k.b. 以下片段距離較近而無法分辨;而2% 膠體搭配「100bp marker」,才可以分辨出100-1000 b.p.。
Gene Cloning | 如何將目標基因克隆到載體中 (下)

.png)